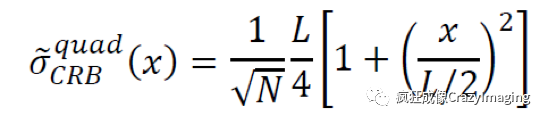2017年,诺贝尔奖获得者Stefan W. Hell在Science上发表了他的又一个突破性技术——2nm定位精度的光学显纳镜(nanoscopy)——MINFLUX(minimal photon flux)1。随后又有两篇应用性文章分别发表在2018年的PNAS和今年的Nature Method上,文章分别展示了使用MINFLUX技术快速追踪单分子跳跃和纳米级3D超高分辨率以及多色超高分辨率图像。这标志着纳米级分辨率显微镜已经开始正式走入生命科学研究领域。在2月24日“病毒与超分辨荧光显微镜”(点击阅读)的文章里,我们和大家交流过显微镜的分辨率和超高分辨率显微镜的概念及种类。MINFLUX处于分辨率的什么位置呢?我们可以参考图中橙色向下箭头所覆盖的区域。从图中可以看出其分辨率已经完全进入纳米量级,称之为显纳镜实不为过。
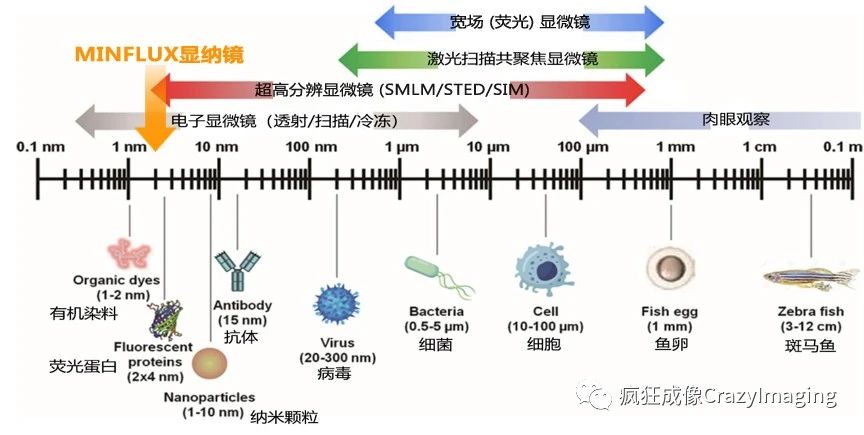
图1 生命体和成像设备分辨率之间的对应关系
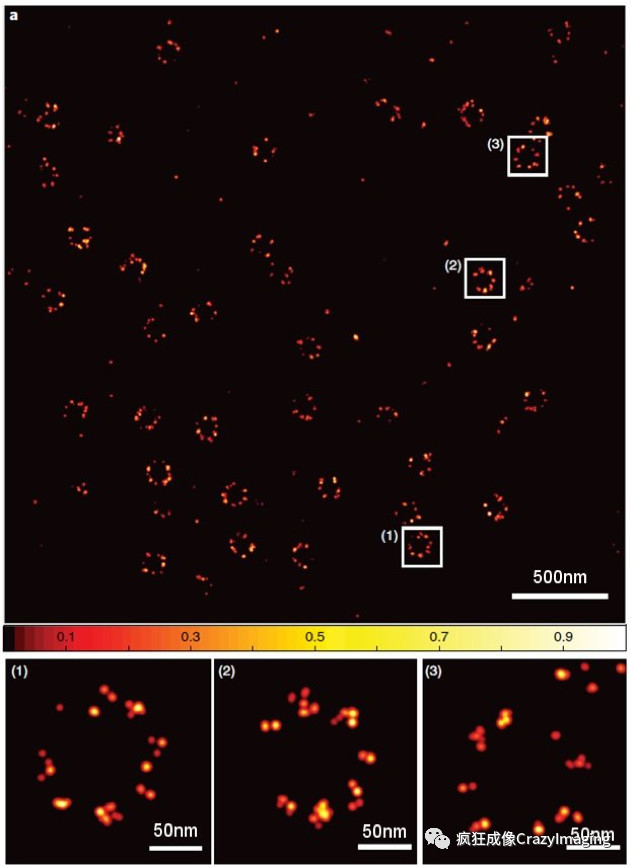
下面我们将根据已发表的MINFLUX文章与大家分析一下这项突破性的技术。首先,我们通过对核孔复合物蛋白和神经元轴突的血影蛋白(spectrin)的成像,给大家展示一下MINFLUX能达到的惊人的分辨率。图2是MINFLUX对U-2 OS细胞中的核孔复合物蛋白Nup96的成像结果2。其中的每一个亮点都是一个被Alexa Fluor 647染料标记了的蛋白亚基。
图2 MINFLUX对U-2 OS细胞的Nup96核孔复合物的成像结果
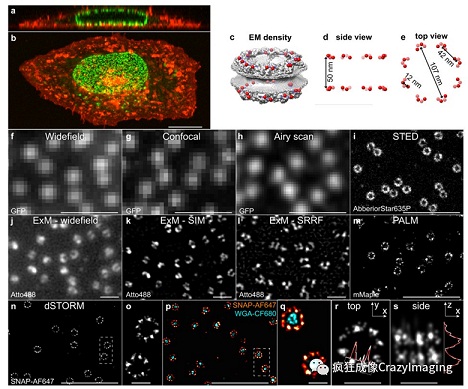
使用Alexa Fluor 647标记Nup96–SNA。可以清晰看到组成核孔复合物亚基分布情况。(1)-(3)为图片a中3个单个核孔的放大。每个点显示的定位信息代表通过其荧光标记表示的单个蛋白质亚基。其他显微镜成像的结果如何呢?请看下图312。该图使用的样品依然研究的是核孔蛋白Nup96。其中,使用最佳状态显微镜能够得到的单分子定位显微镜的最好结果如图m-s所示,它已经是这些图里分辨率最高的了,但与电镜拍照的复合物结果仍然差距很大,只能“显微,而不能显纳”。对比图2中的MINFLUX结果,我们可以看到MINFLUX还能更进一步帮助研究者观察到每一个复合蛋白聚集上实际还有大约2-4个小亚基的结构,这与电镜拍照的纳米级分辨率结果极为一致。对比电镜,MINFLUX可以对活细胞成像。另外其在制样、多色、3D成像等方面的优势也非常明显。因此,我们可以期待这种光学“显纳镜”未来会给我们带来更多突破性的发现。
图3 各种显微镜对Nup-96的拍摄结果
a和b分别是用共聚焦显微镜拍照的Nup96-GFP细胞系的x-z和x-y图像;绿色标记为Nup96-GFP,红色标记的是核膜(DiD)。c为电镜结果,其中红色表示核孔复合物Nup96的 C末端。d和e分别为C末端的侧视图和顶视图。f是Nup96-GFP的宽视野显微镜荧光图像。g为常规共聚焦显微镜的扫描结果。h是使用了Airyscan扫描得到的图像。i是用AberriorStar635P偶联的抗GFP纳米抗体标记的Nup96-GFP的STED图像。j–l都使用了可以膨大样品的expansion microscopy技术,用Atto488偶联的抗GFP纳米抗体标记,不同的是j使用的是宽场显微镜拍照,k和I分别使用了SIM和SRRF成像技术。m使用Nup96-mMaple标记后的PALM成像。n和o是用BG-AF647标记的Nup96-SNAP的dSTORM图像。p和q为用BG-AF647(红色)和WGA-CF680(青色)标记的Nup96-SNAP的双色dSTORM图像。r和s是对核孔复合物做了x,y(r)和z(s)方向的分辨率测量。图中比例尺分别为10μm(b),1μm(f-n,p)和100 nm(o,q,r,s)。
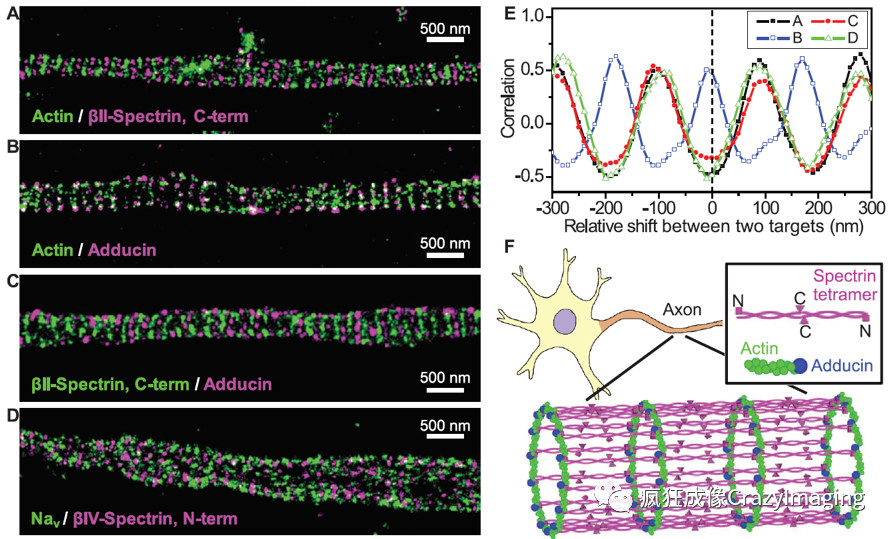
第二个例子关于神经元轴突的血影蛋白(spectrin)。早期的单分子定位的成像结果显示:血影蛋白(spectrin)是如图4中A和F中洋红色示意的那样沿着轴突的方向周期间隔分布的,每个周期长度约为200nm(如图E)8。
图4 单分子定位法获得的血影蛋白和肌动蛋白等在神经轴突上的分布情况最近,北京大学席鹏教授发明了偏振结构光显微技术,并对这一结构进行了观察,结果发现actin虽然呈现了约180nm的周期性排列,但并非首尾相连
(end-to-end),而是呈现了并排结构(side-by-side)13,图5。
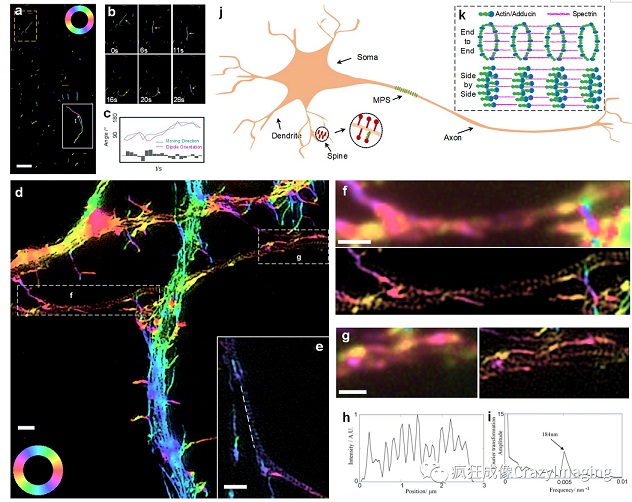
图5 pSIM观测的神经轴突上肌动蛋白分布呈现了平行排列的结果,而非STORM所展示的首尾相连 而在MINFLUX超高显微分辨率下,最新的成像结果如下图6所示。MINFLUX的结果可以更清晰地显示其分布:血影蛋白(spectrin)可能是以螺旋的形式分布的7——是连续分布的,而非STORM成像结果所描述的周期间隔分布。

图6 MINFLUX 拍摄的神经轴突上血影蛋白分布接下来,我们来探讨一下MINFLUX是如何做到这一点的。首先我们问一个问题,这种分辨率,能由其它的超分辨显微镜,比如STED(受激发射损耗)显微镜4和单分子定位显微镜5,6实现吗?理论上来说,是可以的,只要激光功率足够高、荧光基团的稳定性(抗光漂白性)足够好。但现实是,这两点都很难实现11。那为什么MINFLUX可以做到呢?这是因为MINFLUX不是靠检测尽可能高的荧光强度作为目的,而是以检测到尽可能低的荧光强度为目标的。这有什么不同呢?关键就在于泊松噪声,或者称为散粒噪声。泊松噪声是符合泊松分布的噪声,泊松分布用于描述一定时间内少量随机事件的分布。显微成像时,在一个很短的时间窗口内,荧光分子到达检测器的分布就属于泊松分布。因此,在显微图像中,荧光信号总是和泊松噪声如影随形的。反映到图像上,就是图上信号的地方不平滑。泊松噪声有多大呢?从概率上来讲,这个噪声的标准差是信号的平方根。也就是说,荧光强度为100的信号,其噪声的标准差等于10。这就是说,荧光强度测量值为103和98的两个信号从概率上讲是没有区别的。因为这两个信号本身可能是一样的,比如都是100,只是一个有+3的泊松噪声,另一个有-2的泊松噪声而已。所以为了区分N0和N1,我们需要测量值上二者的差值 ΔN > 10(如图7所示)。应用到MINFLUX,如果我们在N0=0的位置进行荧光探测,为了区分N0和N1,只需要探测到 N1=1即可。 也就是说MINFLUX在极低荧光光子数下就能实现,如图73。这也是MINFLUX名字的由来,最小的光流(Minimal Fluxes)1。
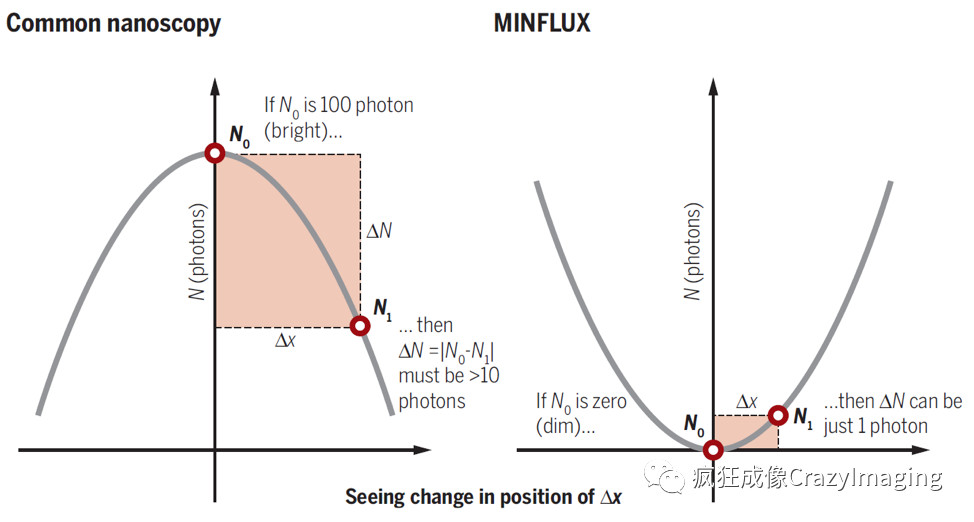
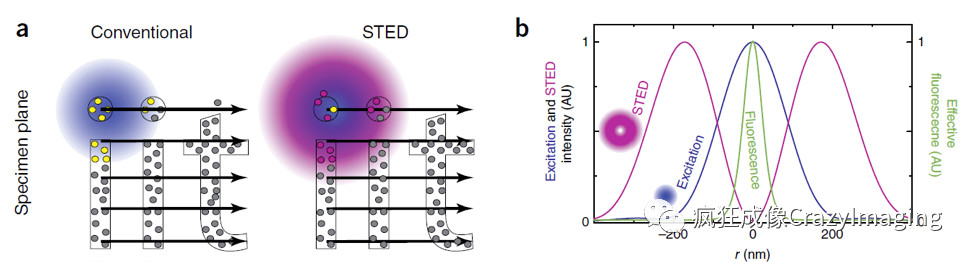
图7 MINFLUX原理示意图科学家是如何实现用最小的光流来定位荧光分子的呢?这就不得不提由Stefan W. Hell发明的另一项技术STED(受激发射损耗)显微镜4和由Eric Betzig与庄小威各自发明的单分子定位显微镜5,6。STED通过在传统的激发光的外围,同时加上一个甜甜圈(donut)状的损耗光,来使得实际激发的区域得以大大减小来实现超分辨成像,如下图8 8。
图8 STED显微镜原理图
而单分子定位显微镜使用随机点亮荧光分子,进而通过高斯拟合定位来实现超分辨,如图9所示6。
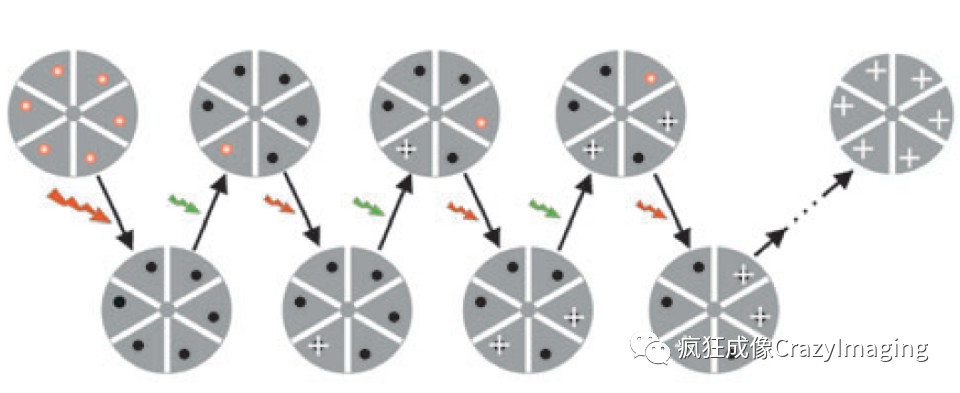
图9 单分子随机定位显微镜原理图
MINFLUX结合了以上两点1。它在很小的区域内随机点亮荧光分子,然后通过甜甜圈状的激光作为激发去精准定位。注意这里的甜甜圈激光和STED的作用完全不同:STED的甜甜圈是用来擦除荧光的,而MINFLUX则是用来激发荧光的。如果甜甜圈激光的中心正好和荧光基团完美重合,这时就检测不到任何荧光;否则就会检测到或多或少的荧光。那MINFLUX是否要经过非常多的迭代才能精确定位到一个荧光分子呢?答案是否定的。因为实际上并不需要真的一步一步地把甜甜圈激光的中心移动到荧光分子的位置,只要迭代精度达到最高统计标准就行了。下面以在一维上定位的例子进行说明。在一维上,实际甜甜圈激光只需要照射两次(移动一次)就可以精确的定位到荧光分子的精确发光位置,如图10所示1。在图10A中,五角星代表一个荧光分子,甜甜圈激光在移动L距离前后各照射了一次荧光分子(绿色粗线),分别得到了荧光强度f0=C*I0和f1=C*I1(C是一个常量)。所以求解该荧光分子的位置Xm,就变成在已知条件f(x)=y、f(X0)=f0和f(X1)=f1下,求解f(Xm)=0。
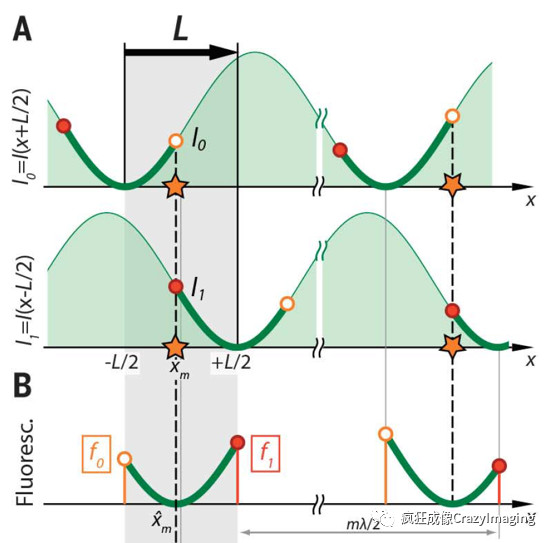
图10 MINFLUX在一维空间的原理示意图
当然,这里的I0、f0、I1、f1都是一个符合泊松分布的测量值。所以解方程得到的位置Xm也符合一定的概率分布,相对真实值有偏差。定位精度可以在数学上计算。从图11中可以看出,在探测到的光子数N一定的情况下,移动距离L越小,最终的定位精度就越高。
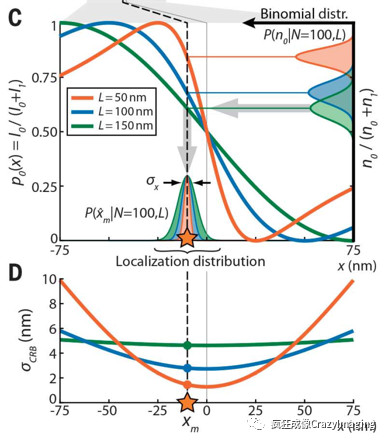
图11 移动距离和定位精度的关系曲线
在数学上,由CR下界(Cremar-Rao Bound, CRB)描述的最高定位精度符合如下公式1。例如,当L=50nm、有100个光子时,最高定位精度是1.3nm。
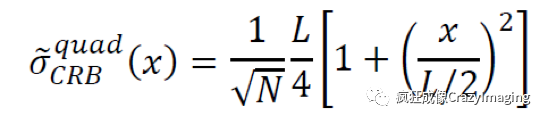
(水印下为L/2)
我们再看看二维上的情况,理论上MINFLUX通过每移动3个位置后得到的荧光强度,就可以来准确的定位单分子荧光,如下图所示1,实际操作中多加一个中心位置以更好的去除背景噪音。
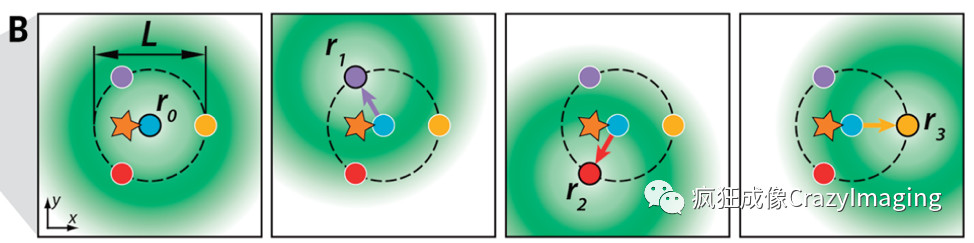
图12 通过移动甜甜圈激发光检测荧光强度变化,来拟合计算二维空间内荧光分子精确定位示意图
而缩小L,能进一步提高分辨率,所以MINFLUX还会通过迭代缩小L,同时提高甜甜圈激光强度来得到更好的定位精度,如下图所示2。
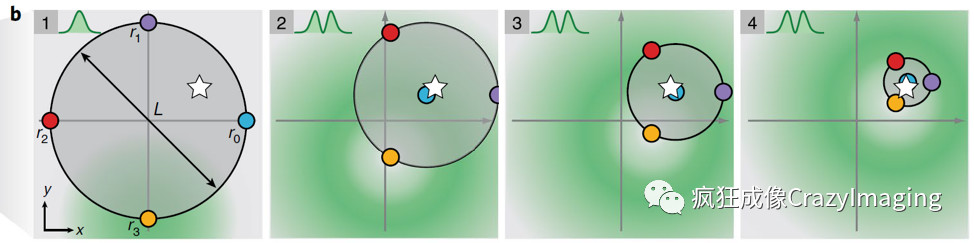
图13通过逐步缩小甜甜圈激发移动距离,来更精确拟合计算二维空间内荧光分子定位示意图
假设MINFLUX和单分子定位显微镜都采集同样多的光子数,我们更可以明显看到两者之间的巨大差异,如图14所示1。这是对DNA折纸的实际MINFLUX成像结果和理想的PALM/STORM模拟成像结果的对比。
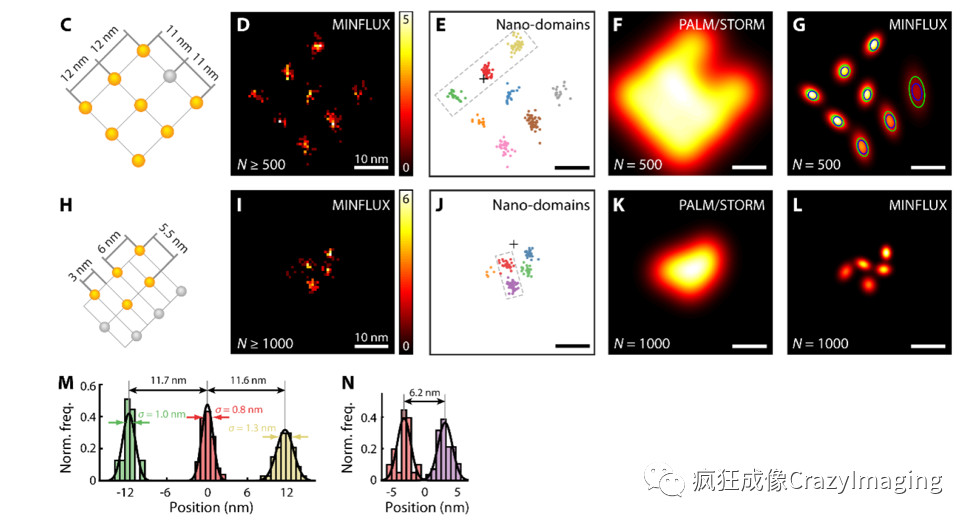
图14 DNA折纸的MINFLUX成像
C:DNA折纸上人工摆放的9个可开关荧光基团,灰色为在整个测量过程中始终保持关暗态的基团。D:MINFLUX直接纳米级定位成像。E MINFLUX定位散点图;虚线框中各颜色区域对应M中的分布。F、G:MINFLUX实拍图的实际双变量正态分布和最佳模拟效果的PALM/STORM理论协方差在相同光子数时的渲染对比。H-L、N:类似于C-G、M,但采用更小的DNA折纸。由于其更高精度的定位能力,MINFLUX展现了根本上超越PALM/STORM的分辨率提高,在室温下以500个信号光子数就达到单纳米级别的分辨率。另一个不同于单分子定位显微镜的是:MINFLUX可以提供非常高的空间、时间分辨率去进行追踪单个荧光分子。图15是使用MINFLUX在活的大肠杆菌内追踪单个核糖体亚基的图像和曲线。它可以实现以20nm的空间分辨率、以28,000位置/秒的时间分辨率去追踪单分子的移动轨迹。
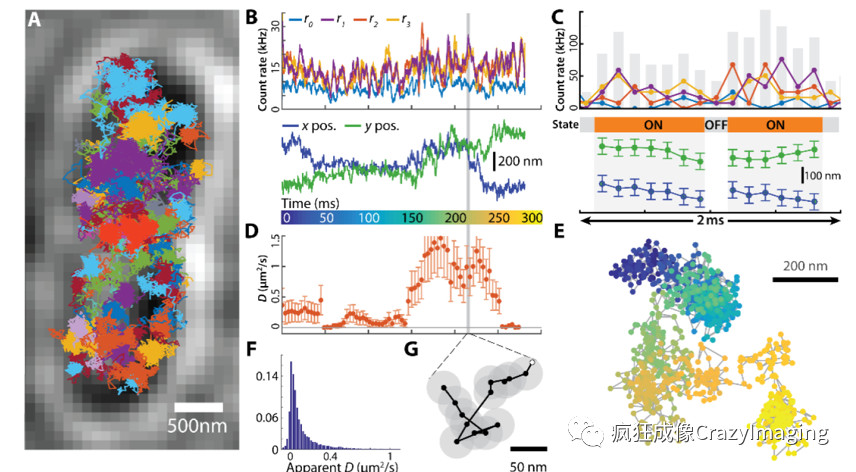
图15 使用MINFLUX在活的大肠杆菌中追踪单分子荧光
跟踪与可切换荧光蛋白mEos2融合的单个30S核糖体蛋白亚基。A图中一共包含有77条独立追踪轨道,背景为细菌的透射图像。B-G为对这些轨迹进行多种分析计算。
鉴于该技术将光学超高分辨率的解析水平史无前例地推向了只有电镜才能达到的纳米水平,我们相信今后一定会有更多的科研成果问世。本文受篇幅所限,如果大家对MINFLUX还想了解更多,可以阅读文末提供的参考文献1,2,10。
参考文献:
1. 2017, Francisco Balzarotti et al., Science.
2. 2020, Klaus C. Gwosch et al., Nature Methods.
3. 2020, Jie Xiao et al., Science.
4. 1994, Stefan W. Hell et al., Optics Letters.
5. 2006, Eric Betzig et al., Science.
6. 2006, Michael J Rust et al., Nature Methods.
7. https://www.abberior-instruments.com/products/minflux/.
8. 2018, Giuseppe Vicidomini et al., Nature Methods.
9. 2013, Ke Xu et al., Science.
10. 2018, Yvan Eilers et al., PNAS.
11. 2018, Yaron M. Sigal et al., Science.
12. 2019, Jervis Vermal Thevathasan et al., Nature Methods.
13. Zhanghao, K. et al. Super-resolution Imaging of Fluorescent Dipoles by Polarized Structured Illumination Microscopy. Nature Communications 2019; 10, 4694.
 最新资讯返回
最新资讯返回