您好,欢迎来到显微镜配件耗材官网!
 最新资讯返回
最新资讯返回从16世纪末荷兰眼镜商人詹森父子发明世界第一台光学显微镜以来,显微镜就成为人们认识微观世界,推动生命科学进步的最主要工具。其后与牛顿同时代的英国著名科学家罗伯特胡克,以及第一个用显微镜观察到细菌等微生物的荷兰科学家列文虎克都对显微镜的制造和应用做出过极其重要的历史贡献。三百多年来随着科技进步,现代的高端宽场荧光显微镜和激光共聚焦荧光显微镜更是可以看清小至单个大细菌(1-5μm左右)和单个细胞(10-100μm左右),甚至亚细胞的尺度(图1),极大推动了现代生物医学的基础研究,做出了卓越贡献。
但是光学显微镜也有局限性,19世纪德国的物理学家阿贝提出了显微镜衍射成像理论,得出了一个重大基本法则,即无论如何提高光学显微镜透镜的制造水平,它的xy平面分辨率(下同)都无法突破一个极限, 这个极限大约为最短可见光---紫光波长的一半,约为200nm左右(紫光波长约为400nm)。即无论如何发展,最好的基于透镜的传统光学显微镜也无法看清分辨出比200nm更小的物体。因此对于尺寸更小的病毒和生物分子,光学显微镜就无能为力了。
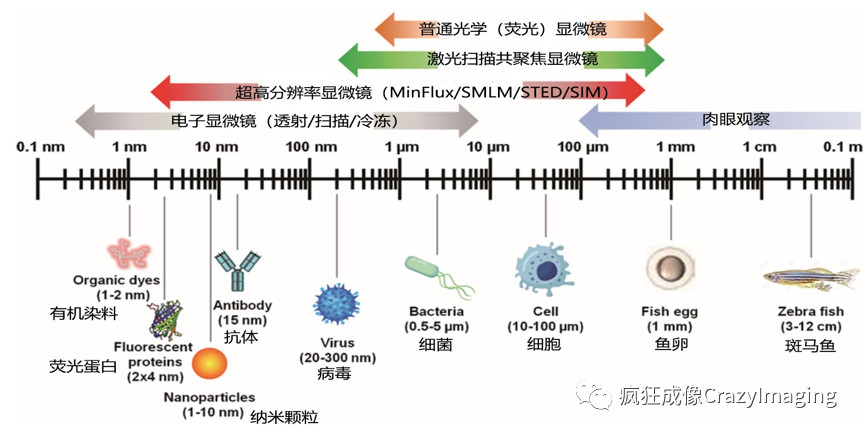
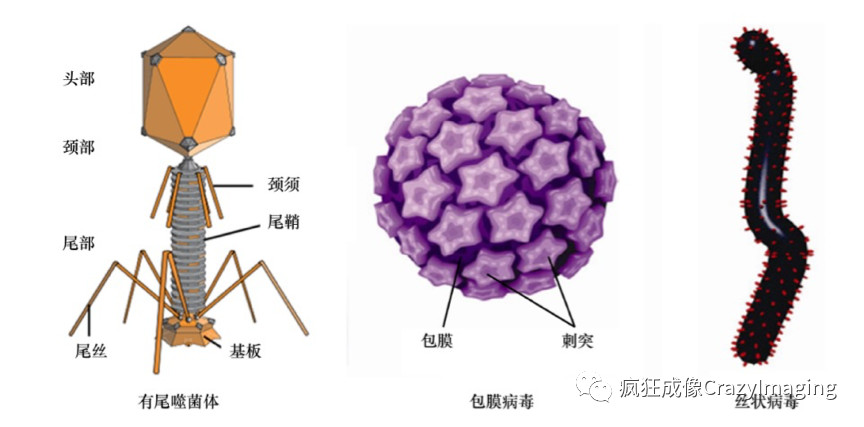 图2病毒是目前已知的地球上最简单的生命单元,不同于一般的细胞生物,它没有完整的细胞结构。通常它是由外壳蛋白和包被在外壳蛋白内的遗传物质(DNA或RNA,统称核酸)两部分组成。由于本身缺乏完整生命过程需要的酶系统及能量转化系统,当病毒游离于环境中时,它只是一个有机大分子,不能长时间存活,只有侵染宿主后才具有生命特征,进行复制传代。它依靠的宿主可以是简单的细菌,比如最简单、最古老的病毒叫噬菌体,它是感染细菌的病毒(图2),也可以感染人类这样复杂的生物体的细胞。病毒感染宿主细胞是一个非常复杂的过程,通俗讲,它必须寄生到一个细胞里面才能实现病毒的“传宗接代”, 如下图,病毒依靠外壳蛋白特异性结合到宿主细胞表面,注入病毒遗传物质到细胞内,利用宿主细胞来大量复制病毒遗传物质并且制造外壳蛋白,在宿主细胞内重新包装成完整病毒,然后破细胞而去进一步感染别的健康细胞,而原宿主细胞死亡。
图2病毒是目前已知的地球上最简单的生命单元,不同于一般的细胞生物,它没有完整的细胞结构。通常它是由外壳蛋白和包被在外壳蛋白内的遗传物质(DNA或RNA,统称核酸)两部分组成。由于本身缺乏完整生命过程需要的酶系统及能量转化系统,当病毒游离于环境中时,它只是一个有机大分子,不能长时间存活,只有侵染宿主后才具有生命特征,进行复制传代。它依靠的宿主可以是简单的细菌,比如最简单、最古老的病毒叫噬菌体,它是感染细菌的病毒(图2),也可以感染人类这样复杂的生物体的细胞。病毒感染宿主细胞是一个非常复杂的过程,通俗讲,它必须寄生到一个细胞里面才能实现病毒的“传宗接代”, 如下图,病毒依靠外壳蛋白特异性结合到宿主细胞表面,注入病毒遗传物质到细胞内,利用宿主细胞来大量复制病毒遗传物质并且制造外壳蛋白,在宿主细胞内重新包装成完整病毒,然后破细胞而去进一步感染别的健康细胞,而原宿主细胞死亡。
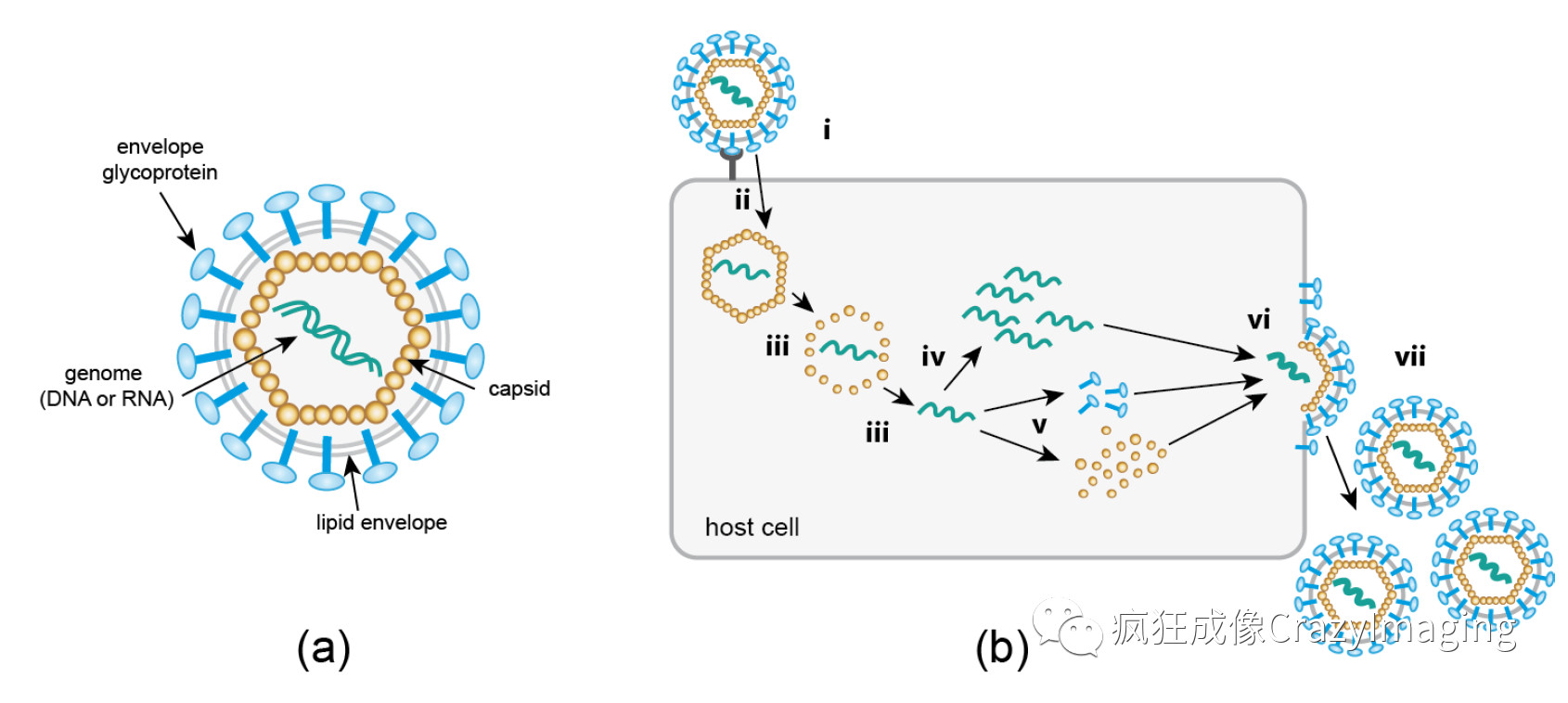
图3
人类对病毒的认知却远远晚于并且小于对细胞和其它微生物(如细菌),最主要的原因是病毒太小了。和正常细胞(10-100μm)或细菌(几微米)相比,一般病毒在100nm 左右(从二十到几百纳米不等),和它感染的人类细胞相比,相当于北京鸟巢体育场里站着的一个人。凭借普通光学显微镜根本看不见它,虽然它广泛存在着。十九世纪末,俄国科学家伊万诺夫斯基就在研究烟草花叶病时发现了病毒的存在,但 “病毒”的形态结构却要等到20世纪30年代后随着电子显微镜的发明才被人类所逐步认识。进入到20世纪30年代,人们对更小的微观世界的追求促进了电镜的发明。德国科学家Ernst Ruska(1986年度诺贝尔物理学奖获得者)发明了透射电子显微镜,利用加速电压产生的电子波长远小于可见光的波长,从而使电子显微镜能达到远超于普通光学显微镜2-3个数量级的分辨率。电子的德布罗意波长由加速电压决定,加速电压越大,电子波长越短,目前最高分辨率的透射电镜可达到0.1nm的分辨率,与可见光显微镜分辨率约200nm的极限相比,其分辨率提高了约2000倍。因此通过电子显微镜人们发现了许多以往光学显微镜看不到的生物结构,如病毒(20-200nm,平均100nm左右),小的细胞器(如核糖体、高尔基体等),大分子(蛋白质、DNA/RNA、从几个纳米到几十个纳米),这也让电镜成为病毒形态学研究最主要的工具。目前肆虐神州,令我们深恶痛绝的新型冠状病毒的样子就是从电镜中看到的直径大约100nm花冠样的结构(如图4)。
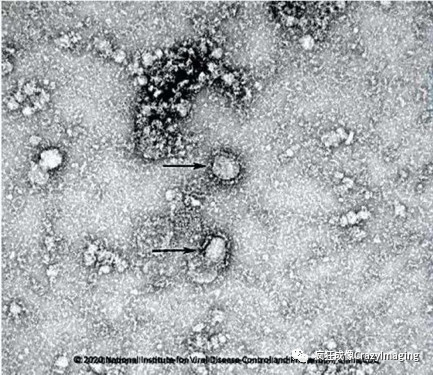
图4(图片来自国家病原微生物资源库)
和别的技术一样传统电镜也有它的局限性,由于传统电镜产生的电子散射对于蛋白质和水没有太大的差异,这就需要在研究细胞时,对样品进行脱水和样品染色等处理,用来增加衬度(对比度),这种处理会损伤样品原来的生物学性状。同时为了避免对电子束的干扰,常规的电镜观测样本必须在真空中,这些因素导致电子显微镜难以研究活的生物细胞。另外,透射电子显微镜观测到的是三维物体的二维投影,不能获得样品内部的三维结构。
为了克服传统生物样品应用于电镜时的制样损伤,上个世纪70年代,科学家发明了冷冻电镜。这一样品制备技术将含有生物大分子的原溶液迅速冷冻于零下196℃的液氮中,减少冰晶形成,保有其生物性状的同时进而降低高能电子对生物标本辐照损伤。美国的A. C. Steven实验室和英国的R. A. Crowther实验室于1997年在Nature同期发表了文献,第一次报告了用冷冻电镜观察到的病毒的亚纳米结构(图5)。最近十多年冷冻电镜技术屡有重大突破,其中三维生物冷冻电镜技术成为研究蛋白及病毒结构、分布与功能的重要手段。该技术的发明人也因此获得2017年诺贝尔化学奖。
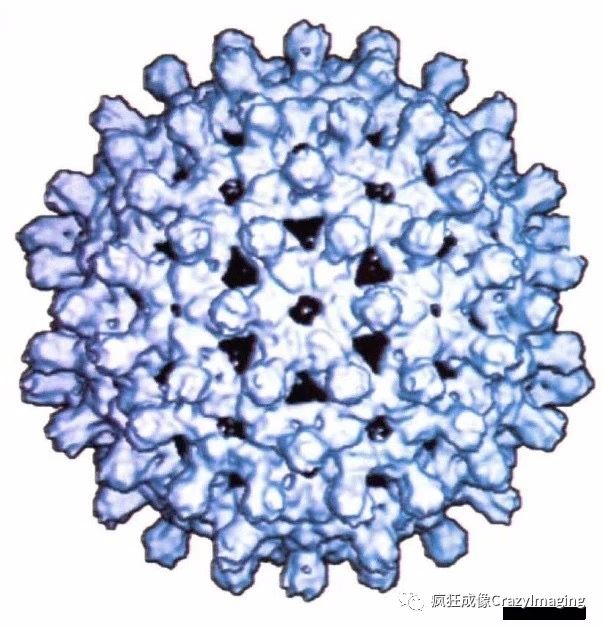
图5(原片来自A. C. Steven实验室于1997年发表在Nature的乙肝病毒,标尺5nm)
可以讲,无论是传统电镜还是最近火爆的冷冻电镜,由于分辨率优势,都对深入解析病毒的结构起到了极其重要的作用。但由于工作原理,电镜使用还存在以下限制:1.传统电镜需要对生物样品进行固定、包埋、切片以及重金属染色等繁琐的过程,标本必须是超薄固定。2.由于电子有限的穿透深度,无论哪种电镜,都不能用于较厚的样品成像(不到1μm)。3.电镜成像实际上只是对附着于生物大分子表面的重金属染料成像,这种重金属染料没有生物特异性标记能力,所以无法区分结构功能不同的生物分子。4.由于制样限制,无论哪种电镜,都无法获取生物过程的动态信息。
从人类与病毒作斗争的漫长历史来看,人类研究病毒最主要的目的还是寻找攻克病毒造成疾病的方法,这就必须深入探究病毒感染宿主的详细机制。也就是除了研究病毒自身结构功能外,必须进一步研究病毒与感染细胞之间的动态过程,哪怕这种动态过程是以静态连续展现出来的。
正因如此,科学家在使用电子显微镜研究病毒几十年后,又将目光重新放回操作简单方便易用的光学显微镜身上。我们知道,之所以光学显微镜不受病毒学研究青睐,最主要的原因就是光学显微镜先天的分辨率低(无法直接分辨200nm以下的目标),从而无法满足对病毒(通常小于100nm)的成像,更不要说直接观察病毒在活细胞内的感染,复制和释放过程了。而现在,由于技术的进步和创新,这一问题可以通过荧光标记(见下)和超分辨率荧光显微镜(见六)来有效的解决。
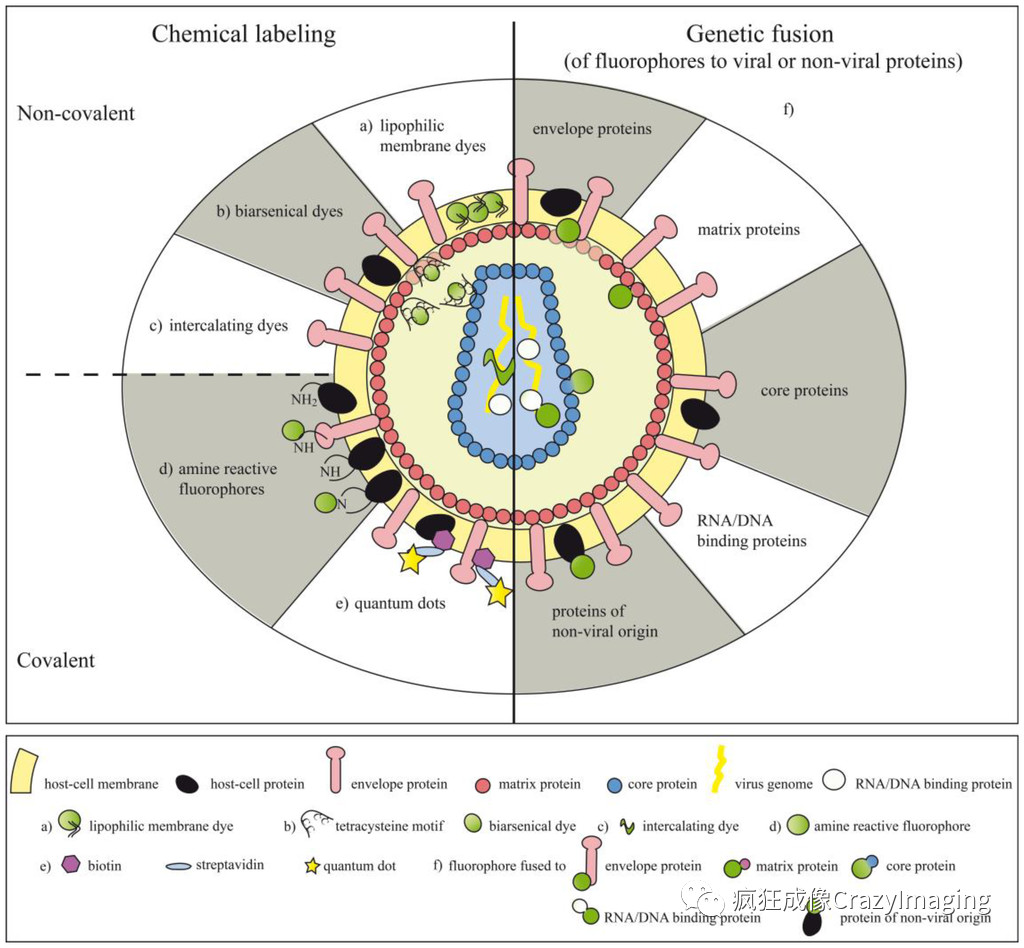
荧光标记是将病毒标记上荧光分子之后,观察可被光学显微镜分辨出的萤光团,利用荧光标记物的放大作用,来间接地观察我们看不见的病毒这样就能使病毒的感染过程可视化,就能够动态追踪病毒在细胞或宿主体内的复制、扩散等生命活动。图6展示了多种利用荧光基团来标记病毒的策略。
从上世纪末以来,以绿色荧光蛋白GFP为代表的多种荧光蛋白陆续实用化,各种新型荧光染料标记物(肽/蛋白标签、有机染料、量子点以及荧光素酶)的产生, 和以共聚焦显微镜为代表的各种高端荧光显微镜的广泛使用,使得对目标中不同的分子进行特异性多色标记(图6),从而实时动态观察病毒感染宿主细胞过程变为可能,极大的改变了传统病毒研究中电镜一家独大的局面,补充了许多电镜手段无法观测到的生物医学内涵。
下面给出几个例子:
如图7,我们可以看到艾滋病病毒HIV-1(eGFP标记,图中亮点)感染活细胞25小时后在细胞中进行病毒组装的分布图(左图,宽场荧光显微镜;右图,全内反射荧光显微镜)。尽管我们在这里使用的荧光显微镜受限于分辨率极限,仍无法识别单个病毒颗粒,但是我们可以通过荧光标记物(染料或荧光蛋白)来特异性标记病毒中的某种结构蛋白,从显示荧光标记物的亮点/亮区(如图)得到宏观聚集的病毒团的分布和动态信息。
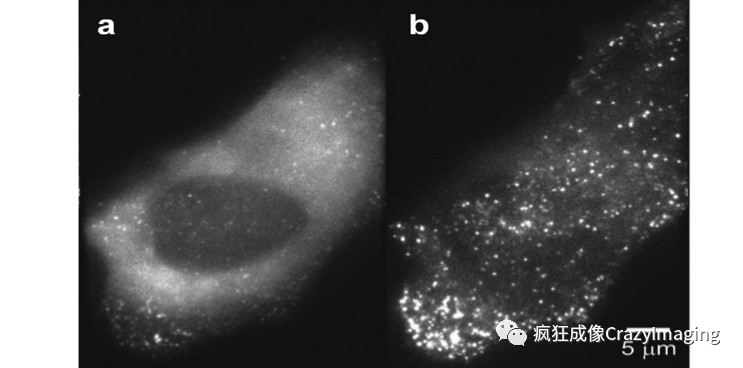
图7
图8中,用间接免疫荧光方法和共聚焦显微镜观察到,绿色荧光标记了其结构蛋白的SARS病毒团与受感染细胞中的核蛋白(用红色荧光标记)在细胞核区共定位的情况。
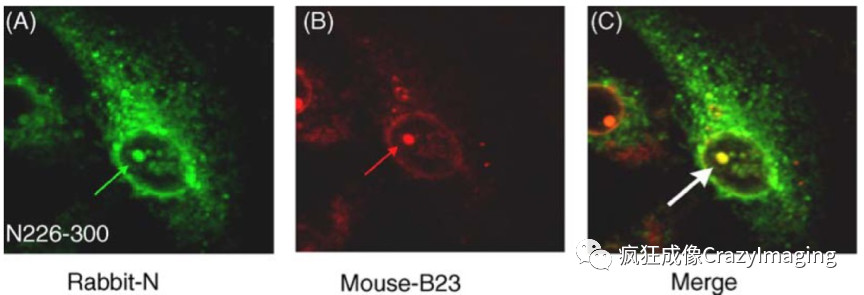
图9是艾滋病HIV-1病毒感染免疫T细胞的动态过程.绿色小点是HIV1的结构蛋白Gag-GFP,用来示踪病毒团从一个受感染细胞(绿色)侵入另一个健康细胞(红色)的过程。
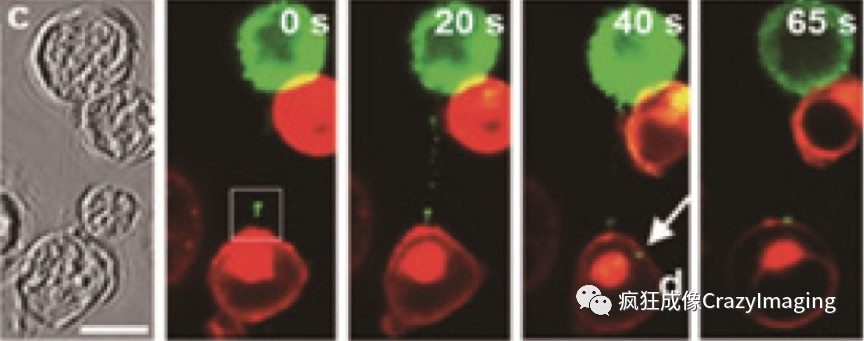 图9
图9无论是最普通的光学显微镜,高端宽场荧光显微镜还是激光共聚焦荧光显微镜,受制于我们前面介绍的阿贝衍射定律,存在一个光学的分辨率极限,无法像电镜一样大幅度提高其分辨率。但能否另辟蹊径来提高光学显微镜的分辨率呢?从上世纪的八十年代以来,科学家们尝试了不同的方法,利用荧光发光的某些机制,规避绕开了传统衍射极限限制,从而在光学显微镜上最终实现了超越200nm极限的分辨率。
真正的突破要从1994年德国马克斯普朗克研究所Stefan W. Hell发明STED超高分辨率方法开始。从那以后的20多年里,各国科学家发明了一系列打破光学衍射极限的荧光超分辨技术,可以使荧光显微镜的分辨率提高2~10倍,目前最高可以在三维上提高两个数量级即百倍(MINFLUX)。为在纳米尺度上三维动态的研究亚细胞结构功能病毒机理,生物大分子作用等,提供了的前所未有的有效手段。因此包括Stefan W. Hell在内的三位对超分辨荧光显微镜研发做出杰出贡献的科学家被授予2014年诺贝尔化学奖。
目前,超分辨显微镜技术可分为三大类(见图10):
第一类是基于单分子定位的显微成像技术(Single Molecule Localization Microscopy,SMLM,例如PALM/STORM)。此前分辨率极限的根本原因是在衍射极限范围内,荧光基团同时发光,相互位置又过于靠近,从而无法被传统光学显微镜同时分辨。因此如果能在时间和空间上隔开记录这些光点,分开辨识,就可以随后组合出超分辨率的图像。此类方法通过一些光学技巧例如光控开关荧光分子,可以实现短时间内只有极少量甚至单个荧光分子发光并被探测到,相机记录下其位置,此过程重复成千上万次,记录下千千万万个来自样品的发光点,然后再利用数学定位算法重组出高分辨图像,可以说是一种以牺牲时间换取空间分辨率的方法(图10A)。
第二类是以受激发射损耗显微镜(STimulated Emission Depletion microscopy, STED)为代表的模式光照明成像方法。这种技术在正常用于荧光激发的衍射极限激光圆斑上,重叠一个同心中空的特殊激光环(即损耗激光环),来同步照射荧光染色样品,图10B。处于圆心部分的样品荧光基团由于没有损耗激光环重叠,可以正常被激发并发射荧光。周边重叠部分的荧光基团被正常激发后,在正常产生荧光之前,由于损耗激光环的存在而迅速从激发态被强行拉回到基态,从而剥夺正常的荧光发射能力,形成一个环形暗区。损耗激光环的强度越大,形成的暗区越大,这种组合方式可以使得圆心处的有效荧光分辨率大大低于衍射极限,实现极高的分辨率。
第三类是结构光照明技术(Structure Illumination Microscopy, SIM)。通过在照射样品时,按特定方式引入含有特定频谱信息的结构化照明遮罩,来采集图像,之后利用相符合的数学算法解析出分辨率低于衍射极限的样品图像 (图10C)。
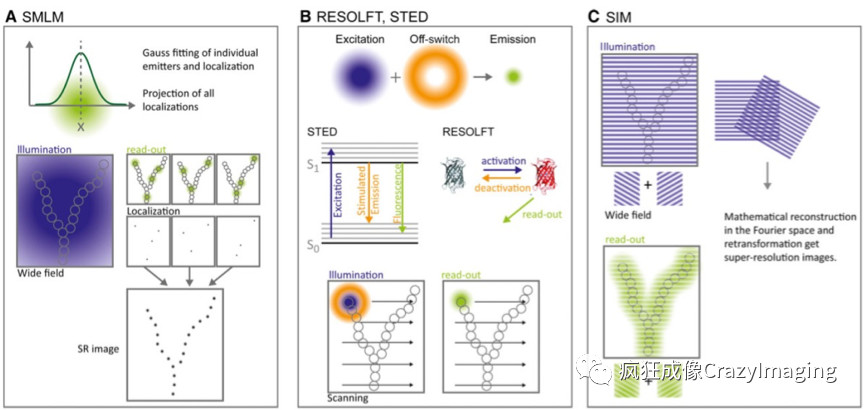
图10
目前商品化的单分子定位和STED的方法最高可以实现20nm分辨率水平。商品化的结构光照明技术能实现的分辨率通常不能突破100nm。因此对于平均尺寸在100nm左右的病毒研究来讲,与共聚焦显微镜(一般220nm分辨率)相比结构光照明技术优势并不明显,所以目前我们能看到用于超分辨研究病毒的文献主要还是集中在前两种方法,这两种方法里显然STED方法更有优势, 因为单分子定位技术整体出图时间较长,不适合研究病毒感染细胞及复制、组装、释放等动态过程的研究。(图11中为自适应照明 MINFIELD STED观察到的HIV-1病毒颗粒)
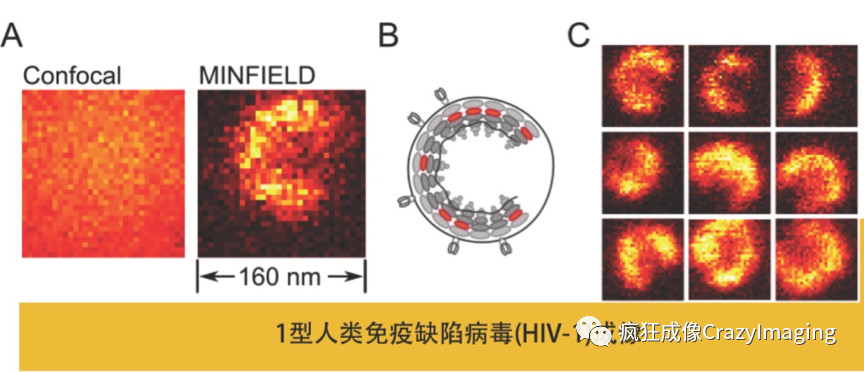
图11 MINFIELD STED 超高分辨率与共聚焦下HIV-1艾滋病病毒颗粒
图12反应了不同显微观察方法下单独的HIV-1病毒颗粒。(A)电镜图可清晰看到静态的病毒形态,包括衣壳(蛋白)包裹着电子致密区(核相关蛋白位置),但病毒表面的Env蛋白无法特异显示出来。 (B)HIV-1 病毒颗粒的假想图。(C)共聚焦模式下的HIV-1病毒颗粒表面的Env蛋白(红色,比较模糊)。(D)与(E)显示同样的病毒颗粒在超分辨模式(大约40nm分辨率水平)下观察,可清晰地展示Env蛋白(红色)在病毒颗粒表面的分布,绿色背景是病毒核相关蛋白Vpr(标尺: A-B为50nm, C-E为100nm)。
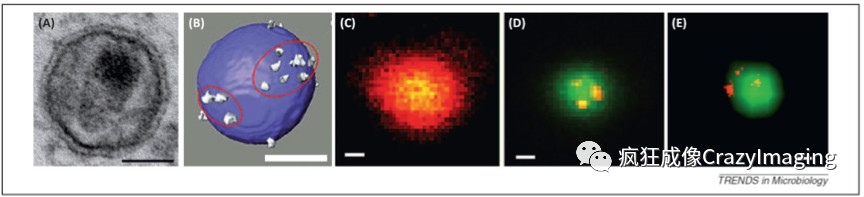
图12
HIV-1病毒尺寸约为直径140nm的球形,其本身已经小于光学衍射分辨率极限,更不用说其表面的蛋白。从图13(标尺均为100nm)中我们可清晰地看出STED超分辨方法采集的HIV病毒表面Env蛋白的分布及数目。而共聚焦显微镜对同种蛋白成像受制于分辨率限制,完全不能解析出同样的生物学信息,并且而产生尺寸失真(任何小于分辨率的物体无论大小看上去都是分辨率大小)。
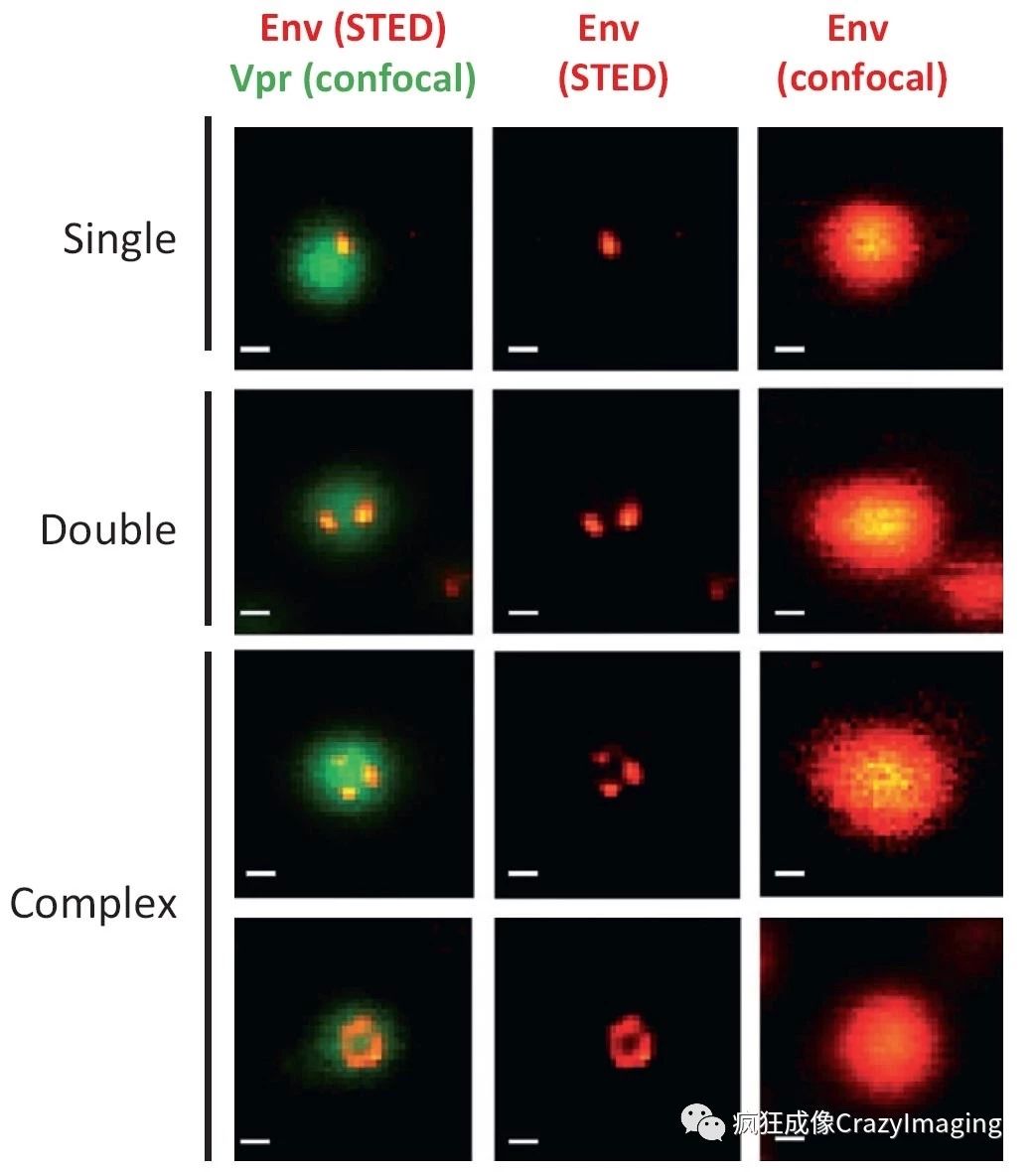
图13
通过上面的分析,我们知道超分辨显微镜在病毒研究邻域应用广阔。最近几年,诺奖得主Stefan W. Hell的团队在已有基础上将STED技术发展到更高分辨率水平(20nm),更多荧光标记(4色)的活细胞实时成像,并且在三维超分辨率上也有突破 (70x70x70nm)。在全面深入研究病毒感染活细胞的机制,以及病毒与宿主复杂时空相互作用中发挥关键性作用。
令人兴奋的是2017年初,Stefan W. Hell团队在《科学》上报道了MINFLUX超分辨率荧光显微镜,这个技术结合STED光照形式和单分子定位荧光特点,实现对生物标本的观察达到三维上小于2nm的目前最高空间分辨率(图14),已经将光学显微镜的分辨率水平提升到一般透射电镜的水平,是共聚焦显微镜的100倍,成为真正名副其实的“显纳镜”。
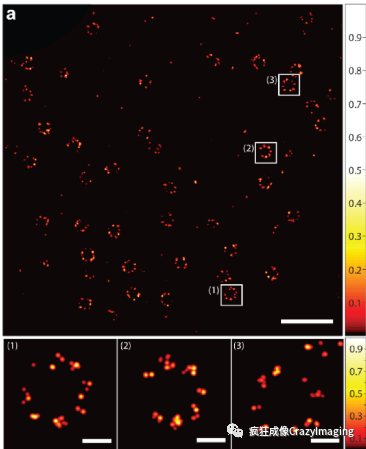
图14(上图1,2,3中标尺为50nm)
我们相信随着该技术商品化以及大规模应用后,“显纳镜”既可以像电镜一样真正“看清”病毒结构,同时又能方便直观的观察到它清晰感染细胞的过程,为科学家找到降伏病毒,造福人类的终极手段。冬天即将过去,春天的脚步已经越来越近,希望人类永远远离病毒的毒害。
参考文献(另部分图片来自网络):
1. Single Viruses on the Fluorescence Microscope:Imaging Molecular Mobility, Interactions and Structure Sheds New Light on Viral Replication Viruses 2018, 10, 250; doi:10.3390/v10050250
2. A stochastic assembly model for Nipah virus revealed by super-resolution microscopy (2018) 9:3050 DOI:10.1038/s41467-018-05480-2 www.nature.com/naturecommunications
3. Multicolor Stimulated Emission Depletion (STED) Microscopy to Generate High-resolution Images of Respiratory Syncytial Virus Particles and Infected Cells (2018) 9:3050 | DOI: 10.1038/s41467-018-05480-2 | www.nature.com/naturecommunications
4. Multicolor Stimulated Emission Depletion (STED) Microscopy to Generate High-resolution Images of Respiratory Syncytial Virus Particles and Infected Cells
Protoc. 2017 September 5; 7(17): . doi:10.21769/BioProtoc.2543
5. Measles Virus Ribonucleoprotein Complexes Rapidly Spread across Well-Differentiated Primary Human Airway Epithelial Cells along F-Actin Rings https://doi.org/10.1128/mBio.02434-19.
6. Visualizing the replicating HSV-1 virus using STED super-resolution microscopy Virology Journal (2016) 13:65
DOI 10.1186/s12985-016-0521-7
7. Teaching an Old Virus New Tricks: A Review on New Approaches to Study Age-Old Questions in Influenza Biology https://doi.org/10.1016/j.jmb.2019.04.038
8. Visualization of early influenza A virus trafficking in human dendritic cells using STED microscopy PLoS ONE 12(6): e0177920. https://doi.org/10.1371/journal.pone.0177920
9. Super-resolved insights into human immunodeficiency virus biology doi:10.1002/1873-3468.12186 Correlative light and electron microscopy methods for the study of virus–cell interactions
doi:10.1002/1873-3468.12153
10. Shedding new light on viruses: super-resolution microscopy for studying human immunodeficiency virus Trends in Microbiology, October 2013, Vol. 21, No. 10
11. Ultrastructural Characterization of Zika Virus Replication Factories http://dx.doi.org/10.1016/j.celrep.2017.02.014
12. Characterization of virus-specific vesicles assembled by West Nile virus non-structural proteins http://dx.doi.org/10.1016/j.virol.2017.03.016
13. Determination of the fold of the core protein of hepatitis B virus by electron cryomicroscopy, Nature, 1997
14. Visualization of a 4-helix bundle in the hepatitis B virus capsid by cryo-electron microscopy, Nature, 1997
15. Fluorosomes: Fluorescent Virus-Like Nanoparticles that Represent a Convenient Tool to Visualize Receptor-Ligand Interactions, Sensors, 2013